Identifican un mecanismo clave para controlar la resistencia a antibióticos y la diversidad genética
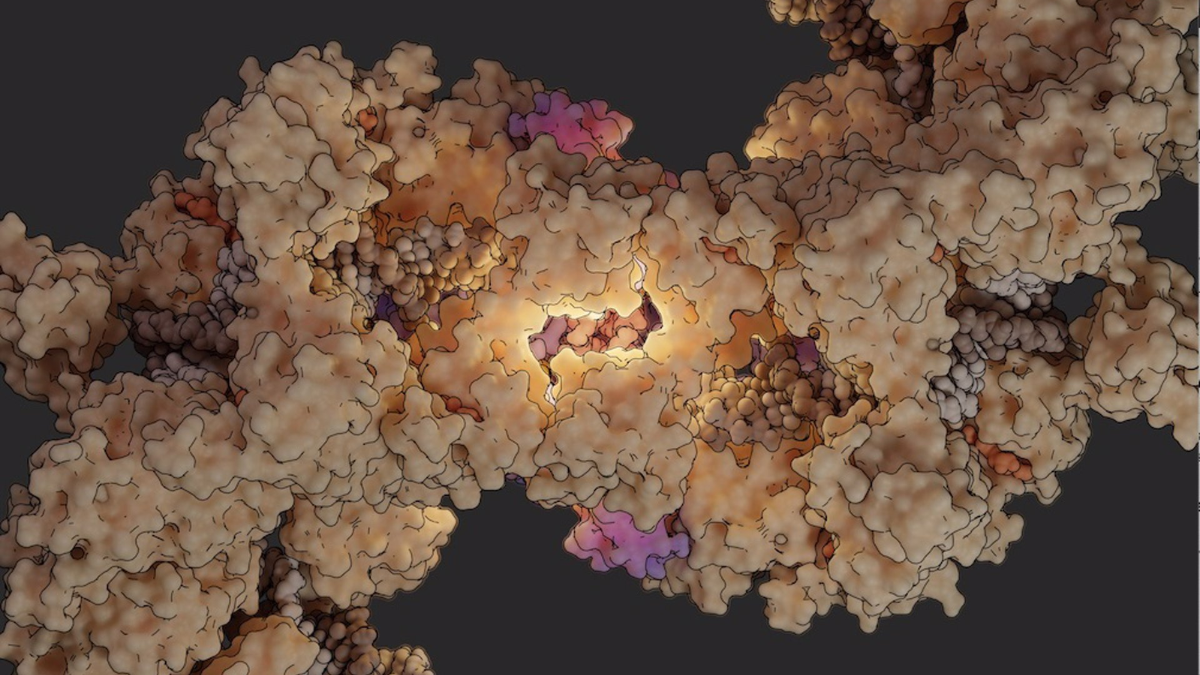
El hallazgo permite entender cómo se regulan las transposasas, unas enzimas esenciales para la transposición del ADN y la transmisión de la información genética
Investigadores del CSIC han descubierto un mecanismo fundamental para regular la diversidad genética y la resistencia a antibióticos en un estudio publicado en la revista Nature.
La investigación arroja luz sobre el mecanismo molecular de la activación de las transposasas, las proteínas responsables de llevar a cabo la movilización de los transposones, uno de los sistemas más abundantes por los que se produce la transmisión horizontal de la información genética.
El estudio, realizado en colaboración con la Universidad Johns Hopkins en EEUU, se ha centrado en la familia de transposones IS21, proporcionando información clave para comprender la regulación de la transposición del ADN.
La revolución de los transposones
En humanos, donde se estima que alrededor del 50% del genoma está formado por transposones o secuencias derivadas de ellos, se han visto relacionados con procesos como la plasticidad neuronal y enfermedades como la esquizofrenia o el cáncer. Estos elementos móviles, además, se emplean como herramientas biotecnológicas y están mostrando un gran potencial en edición genética.
Las transposasas, proteínas responsables de la movilización de los transposones, deben estar altamente controladas para evitar roturas de ADN y recombinaciones cromosómicas con consecuencias potencialmente letales. En algunos casos, su actividad está regulada por proteínas presentes en la célula huésped, mientras que en otros existen factores específicos codificados en el propio transposón que controlan esta actividad. Sin embargo, a pesar de que este proceso ha sido objeto de estudio durante décadas, todavía existen grandes interrogantes acerca de su regulación a nivel molecular.
Un avance en la regulación del ADN
El equipo liderado por el investigador Ernesto Arias del Centro de Investigaciones Biológicas Margarita Salas (CSIC), utilizó técnicas avanzadas de criomicroscopía electrónica combinadas con ensayos bioquímicos y funcionales. Demostraron que una proteína de la superfamilia de ATPasas AAA+, llamada IstB, forma un complejo para unir y mantener el ADN diana con una configuración altamente específica.
«Esta estructura es esencial para posicionar correctamente la transposasa en el sitio de inserción. Además, la interacción entre las dos proteínas provoca un gran cambio conformacional en la transposasa que desencadena su activación y la reacción de transposición», ha indicado Álvaro de la Gándara, primer firmante del artículo.
Estos descubrimientos ofrecen nuevas perspectivas sobre cómo los reguladores de la superfamilia AAA+, como los encontrados en sistemas clásicos como Tn7, Mu y transposones asociados CRISPR, controlan la remodelación del ADN y la activación de sus respectivas transposasas.
«Los resultados de esta investigación ofrecen una visión detallada sobre la regulación de las transposasas, una pregunta fundamental que ha intrigado a los científicos durante mucho tiempo. Además de mejorar nuestra comprensión de la transposición del ADN, este trabajo tiene el potencial de impulsar nuevas investigaciones y aplicaciones en campos como la biotecnología, la ingeniería genética y la biomedicina», ha explicado Ernesto Arias, coautor del estudio.
Este trabajo, con amplias ramificaciones para comprender mejor fenómenos como la generación de inestabilidad genómica o la propagación de la resistencia a los antibióticos, subraya la importancia de estudiar los mecanismos moleculares implicados en controlar funciones celulares esenciales.











